作者简介:刘玉洋(1991—),男,山东日照人,硕士研究生,研究方向为药用植物分子生物学,E-mail:yuyang19911026@sina.cn。
根据NCBI公共数据库中的麦冬序列,开发了52对微卫星分子标记,利用开发的分子标记对浙麦冬主产区现有种质资源进行遗传多样性研究。结果表明,浙麦冬各种质资源遗传多样性较低;聚类分析结果显示慈溪的种质资源相对集中,表明其遗传背景单一,不利于浙麦冬产业的可持续性发展。因此,迫切需要引进其他地区的优良种质资源,以进行遗传改良和新品种选育,从而保证浙麦冬产业的健康发展。
麦冬[Ophiopogon japonicus (Thunb) Ker-Gawl]又称为麦门冬、沿阶草、寸冬等, 是百合科沿阶草属多年生植物, 在《神农本草经》中列为上品药材, 块根作为中药使用, 是一种常用滋阴中药, 对肺燥干咳、虚劳咳嗽、津伤口渴、心烦失眠、肠燥便秘等具有很好的疗效[1, 2, 3, 4]。麦冬主产地在浙江、四川、湖北、福建省, 主要品种包括浙麦冬、川麦冬、湖北麦冬、福建麦冬。其中以浙麦冬为最佳, 是著名的“ 浙八味” 之一。麦冬的主要活性成分, 一般有甾体皂苷、高异黄酮类、多糖等[5, 6, 7, 8]。浙麦冬2015年种植面积达262.2 hm2, 衍生开发的参麦注射液、生脉胶囊等产品在2015年销售额达20多亿元。对近几年文献资料研究发现, 有关麦冬的研究大多集中在其药用成分、药理作用方面[9, 10], 种质资源以及遗传多样性方面研究较少[11, 12]。
研究物种遗传多样性, 对了解物种的起源、估算基因资源分布、确定核心种质并保存、预测种源适应性、进行种质资源开发与利用、进行杂交育种的亲本选配等均具有非常重要的意义。分子标记技术在遗传多样性分析中发挥了重要作用[13]。简单重复序列(simple sequence repeats, SSR)又称微卫星DNA, 其核心序列为1~6个碱基的简单串联重复序列, 广泛存在于真核生物基因组的编码区和非编码区, 具有等位基因数量多、数量丰富、遗传共显性、多态性高、重复性好、分析简单等优点[14, 15]。目前该技术已广泛应用于遗传多样性分析、遗传图谱构建、目标基因定位以及指纹图谱绘制等研究[16]。本研究主要对浙江省内不同地区的麦冬种质资源, 利用SSR分子标记技术揭示其遗传多样性, 为麦冬现有生产种质的识别和评价奠定理论基础, 为现有品种遗传改良和优良新品种选育提供科学依据。
2016年4— 5月, 于浙江省麦冬主产区和其他地区采挖浙麦冬、川麦冬和山麦冬种质资源20份(表1)。剪取新鲜叶片置于采样箱, 带回实验室后-20 ℃冰箱内保存, 用于后续提取DNA。PCR仪(9700, ABI公司); 垂直电泳槽(JY-SCZF-A型, 北京君意东方电泳设备有限公司); 台式高速冷冻离心机(5424R, Eppendorf公司); 数控水浴锅(SCG-4, 宁波新芝生物科技股份有限公司); 电泳仪(PowerPac系列, Bio-Rad公司)。SSR引物由生工生物工程(上海)股份有限公司合成; DNA Marker B、10× Taq Buffer、dNTPs和Taq DNA Polymerase均采购于杭州皓丰生物技术有限公司; 丙烯酰胺、TEMED、过硫酸铵、硝酸银、氢氧化钠和四硼酸钠等化学试剂均采购于华东医药股份有限公司。
| 表1 供试麦冬种质资源来源 |
1.2.1 DNA的提取
麦冬叶片中含有较多的多糖、酚等物质, DNA提取参考Doyle等[17]的CTAB法稍作调整, 从单株嫩叶中抽提、纯化每份材料总DNA。
1.2.2 麦冬SSR标记开发
以Ophiopogon japonicas为关键词, 在GenBank (http://www.ncbi.nlm.nih.gov/)中进行检索, 把得到的麦冬序列以.fasta文件格式保存。利用单机版SSR分析程序MISA (MIcroSAtellite)进行SSR位点的统计和分析(MISA软件的参数:二核苷酸重复次数≥ 6次, 三核苷酸重复次数≥ 5次, 四核苷酸重复次数≥ 4次, 五核苷酸重复次数≥ 3次, 六核苷酸重复次数≥ 3次)。所得序列经stackPACK v 2.2去冗余后, 用软件Primer Premier 3.0(PRIMIER Biosoft International, CA, USA)进行引物设计, 主要参数为:引物长度18~25 bp, 避免二级结构, CG含量40%~70%, 退火温度45.0~65.0 ℃, PCR产物长度100~300 bp。
1.2.3 PCR扩增
PCR总反应体系为20 μ L, 含有50 ng DNA、0.4 μ mol· L-1引物对、60 μ mol· L-1 dNTPs、2 mmol· L-1 MgCl2、1 μ L 10× PCR缓冲液及0.5 U Taq DNA聚合酶。所有PCR反应在PCR仪(9700, ABI公司)中进行, 反应程序:94 ℃ 3 min; 94 ℃ 30 s, 各引物退火温度下复性30 s, 72 ℃ 90 s, 30个循环; 72 ℃ 10 min; 10 ℃保存。
1.2.4 聚丙烯酰胺凝胶电泳
在30 mL的6%丙烯酰胺溶液中加入350 μ L的10%过硫酸铵和25 μ L的TEMED, 混合均匀后水平灌胶, 插上梳子, 除气泡后水平放置, 至胶凝固(约30 min左右)。取5 μ L扩增产物与1 μ L 6× 上样缓冲液混合均匀后点样。在1× TBE缓冲液中, 200 V电泳1 h。电泳结束, 用ddH2O将凝胶漂洗5 min后, 放入500 mL 0.1%的AgNO3溶液中染色, 摇床上摇动10 min, 之后在ddH2O中漂洗10 s。将漂洗之后的凝胶放入含有400 mL氢氧化钠溶液(6 g氢氧化钠, 0.076 g四硼酸钠)和500 μ L甲醛的显影液中, 摇床摇动直至凝胶上出现清晰的条带。最后用ddH2O漂洗约2 min, 取出凝胶。
1.2.5 数据分析
在BIO-RAD visadoc 3.0(Bio-RAD, USA)成像系统中扫描凝胶, 使用Quantity One软件依据DNA Marker计算出每个条带的大小, 然后依据SSR重复单元大小进行人工矫正, 确定等位变异大小(bp)及数目, 形成位点等位基因矩阵; 由软件PowerMarker version 3.25[18]进行丰富度(A)和多态信息含量(PIC)等多样性指数计算。
在NCBI上共搜索到麦冬DNA序列545条, 约462 kb。经生物信息学分析发现共有104个SSR位点:其中, 两碱基重复位点28个, 以(AG)n最多, 共24个; 三碱基重复位点39个, 以(AGC)n最多, 共11个; 四碱基重复位点6个; 五碱基重复位点8个; 六碱基重复位点23个。
从发现的104个SSR位点中, 最终设计得到52个麦冬SSR标记。其中, 包含两碱基重复SSR位点的标记12个, 三碱基重复SSR位点的标记29个, 四碱基重复SSR位点的标记4个, 五碱基重复SSR位点的标记1个, 六碱基重复SSR位点的标记3个; 另外, 还有包含两类重复SSR位点的标记3个, 分别是两碱基和三碱基复合位点的标记OJ-ESSR-9, 三碱基和四碱基复合位点的标记OJ-ESSR-23, 以及五碱基和六碱基复合位点的标记OJ-ESSR-16。52个麦冬SSR标记的PCR产物最小为101 bp, 最大为291 bp; 200 bp以内的17个, 其余的35个均为200~300 bp。
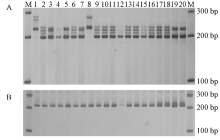
电泳结果表明, 本研究开发的52个麦冬SSR标记中, 有28个可以在20个麦冬种质资源中得到清晰条带, OJ-ESSR-4、OJ-ESSR-13、OJ-ESSR-36和OJ-ESSR-45可以得到1个条带, 其余的24个标记可得到2~7个条带。这28对引物的详细信息见表2。图1是麦冬SSR标记OJ-ESSR-4和OJ-ESSR-38的电泳结果。
| 表2 28个多态性麦冬SSR标记信息 |
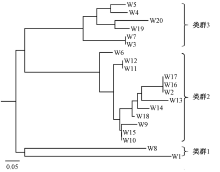
基于24个多态性丰富的SSR标记, 将20个麦冬种质资源聚类成3个类群:类群1, 阔叶山麦冬(W1)和细叶山麦冬(W8)单独聚类成一支, 表明山麦冬和浙麦冬虽然形态上较为相似, 但是他们具有明显不同的遗传背景, 这与山麦冬属于山麦冬属, 与浙麦冬为不同的属的传统分类结论一致; 类群2, 15个浙麦冬主产区种质中的11个和1个衢州种质(W6)聚类成另一个分支, 表明目前浙江省生产上的麦冬种质资源遗传背景相对单一, 这也是由于浙麦冬长期无性繁殖造成的结果; 类群3, 磐安细叶(W3)、萧山浙麦冬(W7)和另外2个浙麦冬(W19和W20)以及2个川麦冬(W4和W5)聚类成第三个分支, 可见浙江省麦冬主产区仍有如天元镇和二塘村那样遗传背景独特的种质, 这有利于浙江省麦冬种质资源的遗传改良(图2)。
本研究发现, 主产区宁波慈溪的浙麦冬相对其他地方的麦冬种质资源, 遗传背景较为单一。这可能会给当地浙麦冬的生产带来潜在的风险, 而且对于麦冬产业的升级也是瓶颈。因此, 为了保证浙江省麦冬产业的健康可持续发展, 迫切需要引进其他地区的优良种质资源, 或者更好地利用现有的如天元镇和二塘村的特异种质, 进行遗传改良和新品种选育。其次, 浙麦冬和川麦冬虽然有效成分组成和含量差异明显, 但在遗传上仍具有一定的相似性, 这为道地药材的分子鉴定带来了一定的难度, 当然具体到浙麦冬和川麦冬有效成分含量的差异是否完全由遗传背景引起还有待进一步研究。第三, 本研究开发的SSR标记可以准确区分山麦冬和浙麦冬, 这些标记可以用于浙麦冬药材的分子鉴定, 打击以次充好, 从而规范药材市场。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|