作者简介:郑炜(1981—),女,山西阳城人,高级农艺师,从事植物检疫方面的工作,E-mail: zhegwei@163.com。
运用PCR直接测序法,对莴苣属植物毒莴苣及其近似种的rDNA ITS区进行序列测定。结果表明,莴苣属植物的ITS序列长度为300~400 bp,设计的引物通用性适用,序列分析可以将属内种间差异分开。
毒莴苣是一种危险性较大的有毒杂草, 不仅严重危害粮食作物的生产安全, 而且易引起人畜中毒, 近年来国内外学者对其形态特征、危害性、生态、生物学特性等进行了系列研究。郭水良等[1]对检疫性杂草毒莴苣的光合特征及其入侵地群落学进行了生态调查; 孙立彦等[2]对山莴苣进行了核型分析研究; 范明松等[3]从山莴苣植株中分离并鉴定出13个化合物, 为医药化合物的开发提供了条件; 王栋等[4]测定了蒙山莴苣根、叶、茎的浸提液对4种草坪杂草种子萌发及幼苗生长的影响; 毕志明等[5]从多裂山莴苣的根中分离鉴定出7种化合物; 梁照文等[6]对毒莴苣及其近似种进行了EST-SSR标记的开发, 构建了11个毒莴苣及其近似种的系统发育树。
DNA分子标记技术为近年来进展最迅速的学科前沿之一, 已被广泛应用于多个物种的种类鉴定。而毒莴苣及其近似种在这方面的研究基本还处于空白。为此, 本研究选取了ITS区作为研究对象, 收集了以毒莴苣为主的几种莴苣属植物, 设计了一对特异性引物, 对其rDNA-ITS区进行了PCR扩增, 并进行了测序验证。
供试材料1号山莴苣、2号毒莴苣、3号莴苣均采自宁波北仑, 4号毒莴苣采自河南新县。
采用高效植物基因组DNA提取试剂盒DNeasy Plant Mini Kit提取DNA, 参照试剂盒说明书进行操作。
根据GenBank中毒莴苣的现有序列, 设计1对引物, 上游引物为ATGCGATACTTGGTGTGAAT, 下游引物为AGTTTCTTTTCCTCCGCTT, 退火温度为53 ℃。
PCR采用TransStart Fastpfu DNA Polymerase 20 μ L反应体系:5× FastPfu Buffer 4 μ L, 2.5 mmol· L-1 dNTPs 2 μ L, Forward Primer 0.8 μ L, Reverse Primer 0.8 μ L, FastPfu Polymerase 0.4 μ L, Template DNA 10 ng, 补ddH2O至20 μ L。PCR仪为ABI GeneAmp® 9700型。
PCR反应参数为95 ℃、5 min为1个循环; 95 ℃ 30 s、55 ℃ 30 s、72 ℃ 45 s共27个循环; 72 ℃ 10 min; 10 ℃保温。PCR扩增结束后, 2%琼脂糖凝胶电泳检测结果。
采用普通琼脂糖凝胶DNA回收试剂盒进行纯化回收, 测序工作在上海凌恩生物科技有限公司的ABI Prism 3730 DNA自动测序仪上完成, 测序引物为CAGAATCCCGTGAACCATCG。
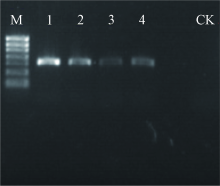
由图1~2可知, 4种莴苣属植物均扩增出有效的目的片段, ITS区序列长度都在300~400 bp, 设计的引物通用性适用, 扩增和测序成功率在100%。
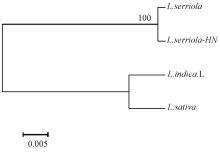
根据测序结果在GenBank中进行序列比对(图3)可知, 山莴苣序列相似度在98%, 莴苣和毒莴苣相似度在99%。根据genedoc软件比对序列, 并mega做进化树(图4)可以看出, 莴苣属内ITS去序列种间的差异较小, 大概在25个碱基左右。从进化树看, 不同采集地的毒莴苣并列成1支, 山莴苣和莴苣自成1支, 种间遗传距离差异较小。
植物DNA序列由于在不同物种、不同分类阶元的系统发育研究中存在保守性差异, 因此针对某一特定的系统学问题选择相适应的分子片段是序列分析中最为关键的一步[7]。rDNA-ITS区序列作为一种基于测序的标记技术已应用于观赏植物和药用植物中。吴玲等[8]对药用植物石蒜属13个种(含变种)的亲缘关系进行了ITS序列分析, 并将其分为三大类; 王东等[9]采用PCR直接测序技术进行山药rDNA ITS区序列的测定, 从分子水平上鉴定和评价道地药材品质性状的变异和品质差异, 以及不同品种之间的亲缘关系等。
本研究通过设计的引物, 比较了4种莴苣属植物的ITS区序列的差异, 由于该区域属内种间差异较小, 种间的差异不很显著, 但试验得到的该属的ITS区序列资料在探讨属内分组、种间关系等分类价值方面具有一定的参考价值, 为莴苣属的种类鉴定和种间亲缘关系提供分子生物学的理论依据。由于本研究得到的样本数量较少, 毒莴苣及其同属内近似种的特异性差异未做鉴定, 有待进一步的研究。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|